Resymé
Et nytt verktøy for identifisering av bakterier
Genet rpoB har et stort potensial til å forbedre identifisering av bakterier. Resultatene fra denne studien viser at sekvensering av rpoB skiller bedre mellom nært beslektede arter enn 16S rRNA-genet.
Om forfatterne:
1. Bioingeniør, MSc og ph.d.-stipendiat ved Mikrobiologisk avdeling, Haukeland universitetssjukehus og Klinisk Institutt 2, Universitet i Bergen.
2. Ph.d., seksjonsoverlege ved molekylærbiologisk seksjon, Mikrobiologisk avdeling, Haukeland universitetssjukehus.
Innledning
16S rRNA-genet har vært brukt som fylogenetisk markør og til identifisering av bakterier siden 1970-tallet. Selv om man i dagens laboratorier hovedsakelig bruker MALDI-TOF (matrix-assisted laser desorption/ionization time-of-flight) til identifisering av bakterier, så finner man jevnlig isolater som ikke lar seg sikkert artsbestemme. I slike tilfeller kan man benytte seg av sekvensering av det bakterielle 16S rRNA-genet. I tillegg brukes amplifikasjon og Sanger-sekvensering av 16S rRNA-genet som dyrkningsuavhengig diagnostikk direkte fra prøvematerialet. Denne direkte 16S-sekvenseringen har noen svakheter, blant annet at genet kan forekomme i mange kopier med variert sekvens. Det kan føre til sekvenseringsresultater som er vanskelige å tolke. I tillegg har 16S rRNA-genet lav mutasjonsrate, noe som gjør det vanskelig å skille mellom mange nært beslektede og klinisk relevante arter.
Det bakterielle rpoB-genet forekommer i kun én kopi per bakteriecelle og har en vesentlig høyere mutasjonsrate, noe som gir bedre muligheter for å skille mellom ulike arter. rpoB har tidligere blitt foreslått som mulig kandidat for identifisering av bakterier, men ingen har klart å designe universelle rpoB-primere. I denne studien har vi utviklet nettopp slike primere, og har undersøkt amplifikasjon og sekvensering av rpoB-genfragmentet som et alternativ til 16S rRNA-genet.
Metode og sentrale funn
Basert på et bredt utvalg av referansegenomer fra NCBI-databasen, designet vi bredspektrede rpoB-primere ved bruk av et såkalt DPO-design (dual-priming oligonucleotide). Denne metoden begrenser kryssreaktivitet mot humant DNA til et lavt nivå, selv om primerne inneholder en rekke degenererte baser.
Vi gjennomførte bred in silico-analyse av det amplifiserte 550 bp lange rpoB-genfragmentet, og analyserte 39 klinisk relevante bakterieslekter. Vi sammenlignet rpoB-genfragmentet med typestammer og andre referanser av høy kvalitet for å vurdere variasjonen mellom arter og genetisk avstand til neste art. Denne analysen viste at rpoB er egnet til å skille mellom nært beslektede bakterier, for eksempel innenfor slektene Streptococcus, Staphylococcus, Enterococcus, Fusobacterium og noen av slektene i Enterobacteriaceae-familien.
Videre sekvenserte vi rpoB- og 16S rRNA-genet fra 115 bakterieisolater (figur 1), som representerte 95 forskjellige arter fra 36 slekter. Bakterieisolatene ble valgt ut på bakgrunn av klinisk relevans eller fordi de er utfordrende å identifisere med 16S rRNA-genet. Alle isolatene ble først dyrket og analysert med MALDI-TOF. Med rpoB klarte vi å identifisere 84% av bakteriene til artsnivå, noe som var betydelig flere enn med 16S rRNA-genet (50%). PCR for rpoB var mindre effektiv enn vår 16S rRNA-PCR, og ga generelt høyere Ct-verdier. Dette hadde imidlertid ingen konsekvenser for identifisering fra bakteriekolonier, der man har rikelig med bakterielt DNA.
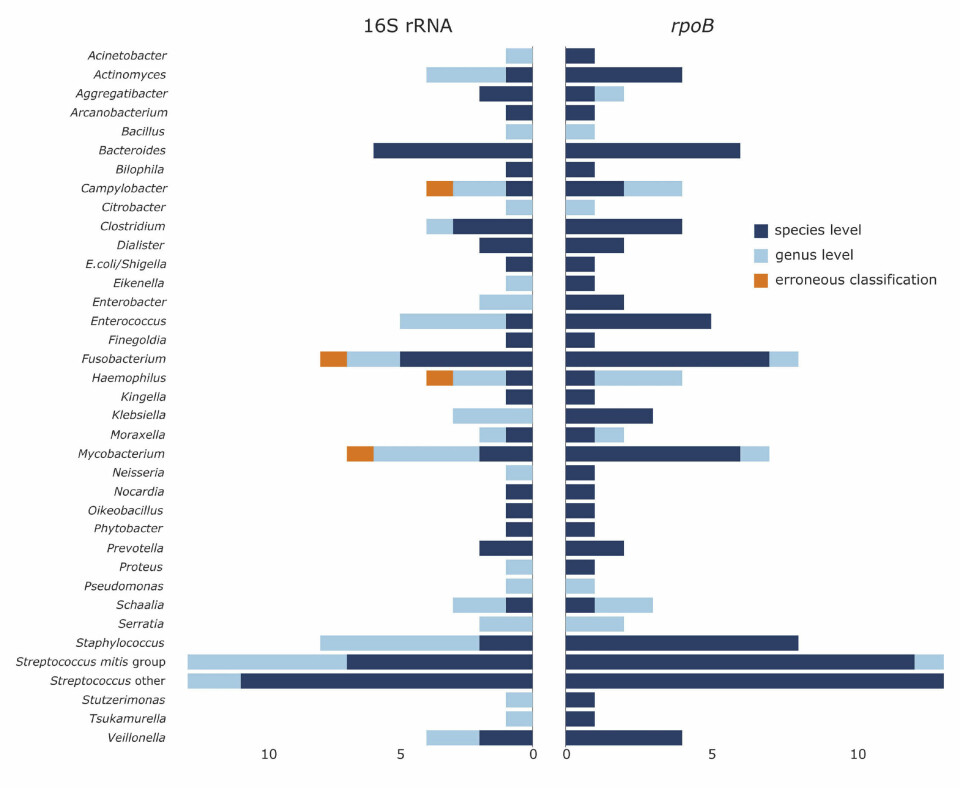
Til slutt evaluerte vi den rpoB-baserte metoden for dyrkningsuavhengig identifisering av bakterier direkte fra prøvemateriale på 33 pasientprøver. Som forventet resulterte den lavere PCR-effektiviteten i en litt lavere sensitivitet, men metoden ga positivt utslag for 88%, og artsidentitet for 91% av disse. Til sammenligning hadde 16S rRNA-metoden en sensitivitet på 100%, men ga artsidentitet bare for 52% av dem.
Konklusjon og betydning for fagfeltet
I denne studien presenterte vi den første universelle PCR-amplifikasjonen av det bakterielle rpoB-genet. Det amplifiserte rpoB-fragmentet gjør det mulig å identifisere mikrober med høyere oppløsning enn 16S rRNA-genet, og metoden kan enkelt implementeres i mikrobiologiske laboratorier. rpoB-metoden egner seg også til bakterieidentifisering direkte fra pasientprøver, men på grunn av en lavere sensitivitet anbefaler vi den som et supplement og ikke som en erstatning for direkte 16S-sekvensering.


























