Denne artikkelen presenterer en retrospektiv gjennomgang av resultater fra MALDI-TOF MS ved St. Olavs hospital, og gir et godt innblikk i bruksområdene for MALDI-TOF MS.
Artikkelen er basert på en bacheloroppgave utført ved Avdeling for Medisinsk Mikrobiologi ved St. Olavs Hospital (AMM).
I løpet av de senere årene har det i større grad blitt tatt i bruk automatiserte identifikasjonsmetoder på mikrobiologiske laboratorier. Metoder for identifisering av bakterier og gjærsopp har ført til en drastisk nedgang i analysetiden.
Et eksempel på en slik analysemetode er Matrix Assisted Laser Desorption Time of Flight Mass Spectrometry (MALDI-TOF MS) (figur 1). MALDI-TOF MS benytter massespektrometri til å detektere molekylmassen til proteinfragmenter hos mikroorganismene. Mikrobenes proteiner fragmenteres og ioniseres, og molekylmassen til frie ioner detekteres i et vakumkammer (figur 1) (1-3). Deretter sammenliknes mikrobens spektrale proteinmønster med en samling av kjente mikroorganismer med etablerte proteinmønstre i en database. Resultater vurderes ut fra likheten mellom bakterieisolatets spektrale proteinmønster og referansespektra i instrumentets database - og angis som en tallverdi (score). Dette muliggjør rask identifikasjon av bakterie- og soppisolater (1, 4).
Det finnes to metoder for opparbeidelse av materiale til identifikasjon ved bruk av MALDI-TOF MS: «Direct Transfer» og proteinekstraksjon (vanligvis maursyreekstraksjon). Metoden «Direct Transfer» innebærer at deler av den aktuelle bakteriekolonien høstes og deponeres på en rustfri stålplate direkte fra en agarskål uten annen forbehandling. Proteinekstraksjon benyttes i de tilfellene man ikke får identifisert ved «Direct Transfer», for eksempel bakterier som har ekstra tykt peptidoglykanlag. Metoden brukes dessuten rutinemessig når gjærsopp identifiseres ved bruk av MALDI-TOF MS (5).
Denne studien er en retrospektiv gjennomgang av resultater fra MALDI-TOF MS over en periode på fire måneder ved Avdeling for Medisinsk Mikrobiologi ved St. Olavs Hospital (AMM). Avdelingen hadde siden anskaffelsen av MALDI-TOF MS, hatt mistanke om at identifiseringsresultatene for bakterieisolater ikke var så gode som diverse studier hadde antydet. Metodene «Direct Transfer» og maursyreekstraksjon ble benyttet for å undersøke om mistanken var begrunnet.
Materiale og metode
Resultater fra alle prøver analysert med MALDI-TOF MS i løpet av en periode på fire måneder, ble hentet ut fra dataarkiverte rekvisisjoner og systematisert i Microsoft Office Excel. Totalt ble det hentet ut informasjon fra 3588 resultater, på henholdsvis 3327 bakterieisolater og 261 gjærsopp. Det ble samlet informasjon om type prøvemateriale, navn på bakterie- eller gjærsoppisolat, scoreverdi, samt eventuelle tilleggstester utført for identifikasjon. Tre kategorier av scoreverdier ble etablert og isolatene ble plassert i aktuell kategori. Et resultat med scoreverdi > 2,0, ble vurdert som sikker identifikasjon på artsnivå, som er høyeste grad av identifikasjon. Isolater med scoreverdi < 2,0 og ≥ 1,7, ble godkjent som identifikasjon på slektsnivå, men dette resultatet måtte bekreftes enten ved hjelp av cellemorfologiske studier eller ved fenotypiske tester av isolatet. Bakterieisolater med scoreverdi < 1,7 ble ikke godkjent for svarutgivelse, og det var i disse tilfellene aktuelt med maursyreekstraksjon og påfølgende reanalyse på MALDI-TOF MS (5).
MALDI biotyper (Bruker Daltonik GmbH, Tyskland) ble benyttet for analyse. Databasen som ble brukt i studien (Biotyper database, version3.0) var oppdatert 27. februar 2013 og inneholdt en samling av litt over 4300 referansespektra i henhold til MALDI-TOF MS loggbok og PC-logg på instrumentet.
Resultater
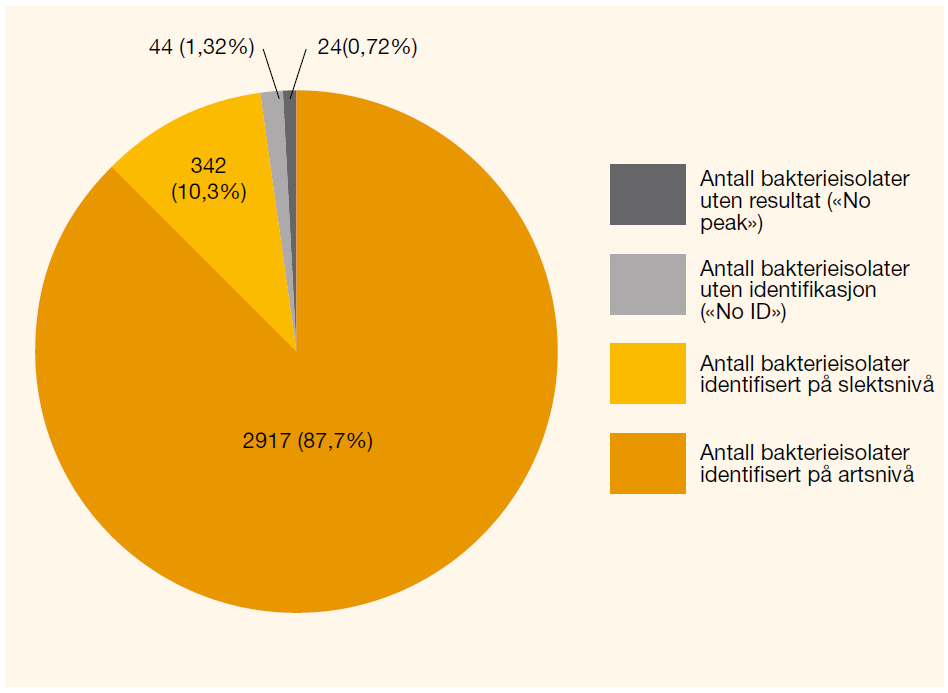
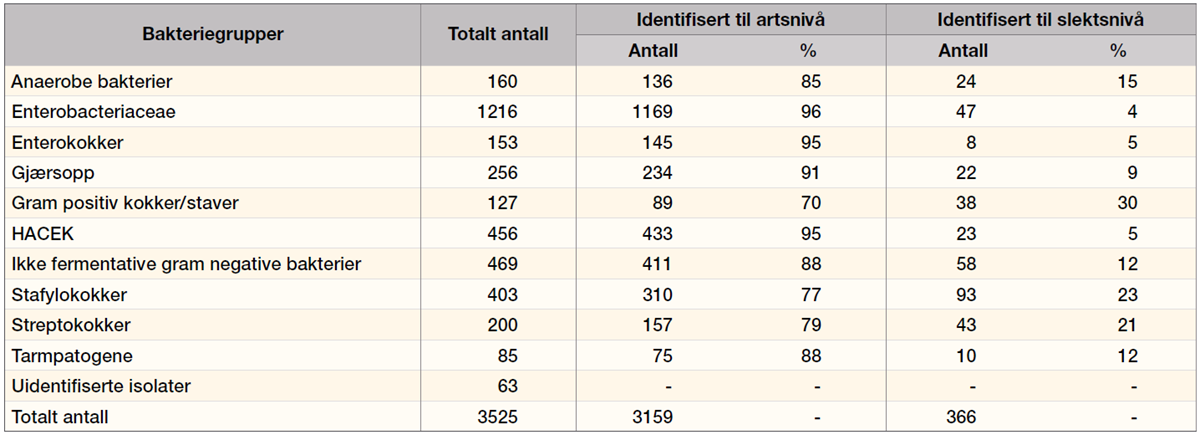
Resultatene fra 3327 bakterieisolater analysert med «Direct transfer» viste at 87,7 % av bakterieisolatene ble identifisert til artsnivå og 10,3 % til slektsnivå, mens 2 % av bakterieisolatene ikke ble identifisert, herunder gruppene «No peak» og «No ID» (figur 2). Gruppene stafylokokker, streptokokker og andre gram positive kokker og staver utgjorde den største andelen (> 70%) av isolater som kun ble identifisert til slektsnivå og ikke artsnivå (tabell 1).
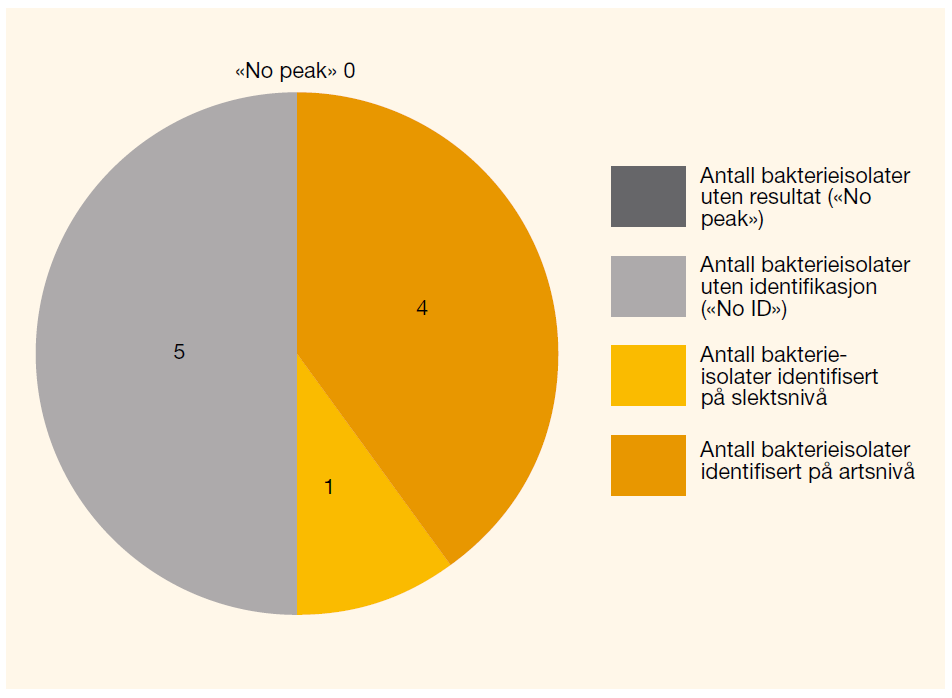
Av de 68 uidentifiserte isolatene som var analysert med «Direct Transfer», ble ti isolater maursyreekstrahert og reanalysert på MALDI-TOF MS. Resultatene viste at metoden identifiserte fire av de ti bakterieisolatene til artsnivå, én til slektsnivå, mens de resterende fem fortsatt ikke ble identifisert (figur 3). I tillegg viste resultatene at alle bakterieisolatene som ble ekstrahert med maursyre fikk topper i massespekteret, da ingen fikk resultatet «No peak».
Totalt ble 87,8 % av bakterieisolatene identifisert til artsnivå og 10,3 % til slektsnivå. 1,9 % fikk ingen identifikasjon, selv etter maursyreekstraksjon og påfølgende reanalyse på MALDI-TOF MS.
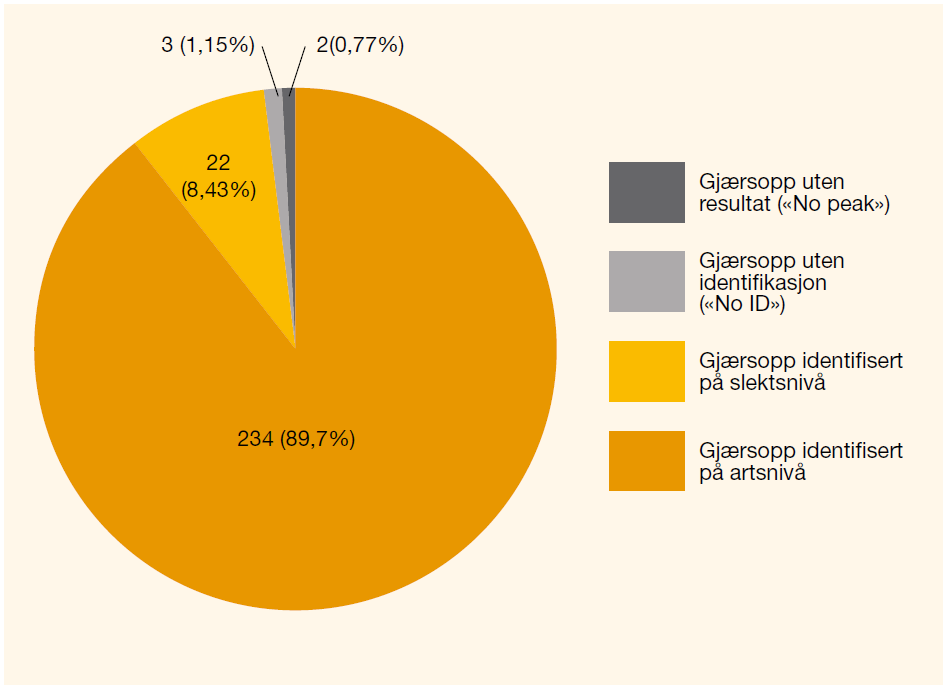
Resultatene av de 261 isolatene innenfor gruppen gjærsopp viste at 89,7 % ble identifisert til artsnivå; 8,4 % til slektsnivå mens 1,9 % ikke ble identifisert (figur 4).
Diskusjon
Denne studien har retrospektivt analysert MALDI-TOF MS score-verdier av bakterie- og soppisolater fra en periode på fire måneder. 87,8 % av bakterieisolatene ble artsidentifisert, 10,3% slektsidentifisert mens 1,9 % fikk ingen identifikasjon. En av årsakene til at 1,9 % av bakterieisolatene ikke ble identifisert kan skyldes at databasen ikke inneholdt referansespektra som samsvarte med bakterie- eller soppisolatenes spektrale mønstre. En annen årsak kan være forbehandlingsfeil av isolatene, som for eksempel at for lite eller for mye prøvemateriale ble avsatt på den rustfrie stålplaten. Feilkilder og tilfeldige feil ved maursyreekstraksjon kan også være en årsak.
Resultatene fra studien er i godt samsvar med liknende studier. I en studie utført av Seng og medarbeidere (6) ble 1660 bakterieisolater analysert med MALDI-TOF MS. Av disse ble 84,1 % identifisert til artsnivå, 11,3 % til slektsnivå og 2,8 % ikke identifisert. Sistnevnte ble forklart med inadekvat databaseoppdatering. I en lignende studie utført av Bizzini A og medarbeidere (7) ble det analysert 1371 bakterieisolater hvor 93,2 % ble identifisert til artsnivå, 5,3 % til slektsnivå mens 1,5 % av bakterieisolatene ikke oppnådde identifikasjon. I den sistnevnte studien var det nødvendig å ekstrahere 25,6 % av de artsidentifiserte bakterieisolatene for å oppnå identifikasjon. I vår studie var det 68 isolater som etter «Direct transfer» ikke ble identifisert, kun ti av disse ble valgt ut for ekstraksjon. Av disse ble fire identifisert til artsnivå etter reanalyse (figur 3). Til sammen ga dette 2921 artsidentifiserte bakterieisolater hvor kun fire var ekstrahert (figur 2 og 3). Selv om denne andelen kunne økt noe dersom alle de 68 uidentifiserte isolatene var blitt ekstrahert, så viser tallene at AMM ikke hadde like stort behov for ekstraksjon for å identifisere isolatene til artsnivå. Dersom AMM hadde rutiner for å ekstrahere bakterier som kun var identifisert til slektsnivå, kunne kanskje andelen av artsidentifiserte bakterieisolater økt ytterligere. Resultatene avkreftet AMMs mistanke om dårlig identifiseringsresultat på MALDI-TOF MS.
Årsaken til at kun ti av 68 bakterieisolater ble sendt til maursyreekstraksjon kan være at AMM heller valgte å sette opp andre identifikasjonstester i stedet for maursyreekstraksjon. Da studien ble utført, fantes det ikke klare retningslinjer i laboratoriets prosedyrer i tilfeller uten resultat etter «Direct Transfer». I slike tilfeller kan det derfor ha blitt et personlig valg av videre identifikasjonsmetode. Dette kan være årsaken til at maursyreekstraksjon ble valgt bort til fordel for tradisjonelle metoder som de ansatte er godt kjent med. Maursyreekstraksjon, som er en relativt tidskrevende prosess, kan også ha blitt valgt bort til fordel for mer tidsbesparende metoder, som for eksempel API (bioMerieux, Frankrike). I denne sammenhengen er tidsbesparelsen sett i forhold til tiden det tar for laboratoriepersonalet å utføre metoden, og ikke tiden det tar å oppnå et resultat. Som vist i figur 3 ble kun 50 % av de maursyreekstraherte bakterieisolatene arts- og slektsidentifisert etter reanalyse, men figuren viser også at alle isolater fikk topper i massespekteret etter ekstraksjon (ingen med resultatet «No peak»). Det indikerer at maursyreekstraksjon har ført til ekstraksjon av proteiner, men at det ikke fantes referansespektra i databasen som samsvarte med isolatets spektrale mønster. Ufullstendig maursyreekstraksjon hvor bakteriens cellevegg bare delvis er ødelagt, kan ha gitt en sammensetning av proteiner som ikke er representativ i forhold til bakteriens forventede proteinsammensetning.
Konklusjon
Studien har vist at AMM ved hjelp av MALDI-TOF MS oppnår en høy prosentandel artsidentifiserte bakterieisolater ved bruk av «Direct-Transfer», sammenlignet med liknende studier. Maursyreekstraksjon er en viktig del av identifiseringen av bakterieisolater som ikke identifiseres ved «Direct-Transfer», og ved identifisering av gjærsopp.
Instrumentet som benyttes til MALDI-TOF MS er kostbart i innkjøp, men reagenskostnadene er svært lave. Dette, sammen med den raske og sikre deteksjonen, gjør MALDI-TOF MS til en meget velegnet analysemetode for rutinediagnostikk i medisinsk mikrobiologi.
Takk
Vi ønsker å rette en stor takk til fagveilederne, overlege Aleksandra Jakovljev og overlege Kåre Bergh ved Avdeling for Medisinsk Mikrobiologi, St. Olavs Hospital, samt de ansatte ved samme avdeling for bistand og hjelp ved utarbeidelse og gjennomføring av bacheloroppgave og artikkel.