FAG i praksis
Revolusjon innen sekvenseringsteknologi: Framgang med sjumilssteg
Artikkelen gir innblikk i den rivende utviklingen innen sekvenseringsteknologien de siste tiårene og hvordan teknologien benyttes innen medisinsk genetikk. Den beskriver også hvordan genetisk utredning foregår, fra første kontakt med helsetjenesten frem til endelig svar.
Av LISS ANNE SOLBERG LAVIK, MARIE LUNDBÆK, KRISTINE MISUND og WENCHE SJURSEN
Framgang med sjumilssteg.pdf(379 KB)
I fagfeltet medisinsk genetikk diagnostiserer man sykdommer som har oppstått enten fordi en genfeil er arvet, eller fordi en genfeil har oppstått spontant. Genetiske feil kan være endringer som påvirker kromosomenes eller genenes struktur eller funksjon, noe som gjør oss mer sårbare for utvikling av sykdom. Genetiske faktorer kan virke alene eller i samspill med miljøfaktorer. Vi antas å ha cirka 21 000 proteinkodende gener, og disse er bare delvis kartlagt med tanke på asso-siasjon til sykdom. I dag kjenner man til mer enn 6000 ulike genetiske tilstander (1) og man finner stadig nye genetiske endringer som kan knyttes til utvikling av sykdommer, noe som ny sekvenseringsteknologi har bidratt vesentlig til. I tillegg til tradisjonelle PCR-baserte-metoder som sangersekvensering av DNA og RNA, og fragmentanalyse av spesifikke genetiske markører, har ny sekvenseringsteknologi ført oss sjumilssteg fremover. Nestegenerasjonssekvensering (NGS) (også kalt andregenerasjonssekvensering) har gjort det mulig å undersøke mange gener eller hele det menneske-lige genom i én enkelt analyse.
Omtrent åtte prosent av alle mennesker utvikler en genetisk sykdom i løpet av livet (2). Ved Avdeling for medisinsk genetikk (AMG) ved St. Olavs hospital benytter vi NGS til diagnostisering av arvelig kreft, syndromer og utviklingsavvik i fosterlivet. Hensikten med denne artikkelen er å beskrive den rivende utviklingen som har vært de siste årene innen gentesting ved hjelp av sekvensering.
Arvelig kreft
Kreft oppstår på grunn av ukontrollert cellevekst. Økt risiko for kreft kan være arvelig, og det er kartlagt mange risikogener innen arvelig kreft. Arvelige kreftsyndromer hvor årsaksgenet er kjent, utgjør cirka fem til ti prosent av alle kreftformer. For eksempel har noen familier økt risiko for bryst- og eggstokk-kreft på grunn av en genfeil i BRCA1- eller BRCA2-genet, og andre familier har økt risiko for tarmkreft på grunn av feil i ett av fire gener involvert i DNA mis-match-reparasjon (Lynch syndrom) (3). Per i dag kjenner vi mer enn 50 ulike arvelige kreftsyndromer og mer enn 80 assosierte gener (4).
Syndromer
Genetiske syndromer er definert som en sammenstilling av symptomer (fysiske tegn og mulige utviklingsavvik) som ofte opptrer sammen, og hvor genetisk årsak er kjent. Årsaken kan være nedarvet eller nyoppstått og omfatte en enkel genetisk variant (for eksempel Duchenne muskelatrofi, med feil i DMD-genet), eller endringer på kromosomnivå (for eksempel Downs syndrom – tre utgaver av kromosom 21). Flere tusen ulike gener involvert i syndromer er kjent, og de klassifiseres ut fra fenotyper.
Fosterdiagnostikk
Det er utviklet NGS-baserte metoder for undersøkelse av kromosomavvik som kan oppstå i fosterstadiet. Ved indika-sjoner på avvik hos fosteret ved ultralyd eller ved høy alder på mor, kan kromosomavvik undersøkes i blodprøve fra mor med NIPT (ikke-invasiv prenatal test, som utføres i samarbeid med Avdeling for medisinsk biokjemi). DNA isolert fra mors blod inneholder en liten andel av sirkulerende DNA fra fosteret, og det er tilstrekkelig til at man kan analysere for trisomi 13, 18 og 21.
Hvorfor er det viktig med gentesting?
Gentesting kan være en målrettet undersøkelse av en kjent genetisk variant som er påvist i en familie, eller det kan være en undersøkelse av ett eller flere gener for å prøve å finne en årsak til sykdom.
Når den sykdomsgivende genetiske varianten i en familie er identifisert, er det mulig å tilby gentesting av slektninger. Ved å identifisere bærere av denne varianten, kan disse tilbys oppfølging fra helsetjenesten i form av regelmessige undersøkelser og kontroller som for eksempel MR (magnetisk resonans) og CT (computertomografi). På denne måten kan man oppdage for eksempel kreftutvikling i et tidlig stadium og dermed forhindre videre utvikling og død. Figur 1 gir en skjematisk oversikt over genetisk utredning for arvelig kreft ved vår avdeling, og faktaboksen «Genetisk utredning» gir en forklaring på aktuelle begreper ved gentesting.
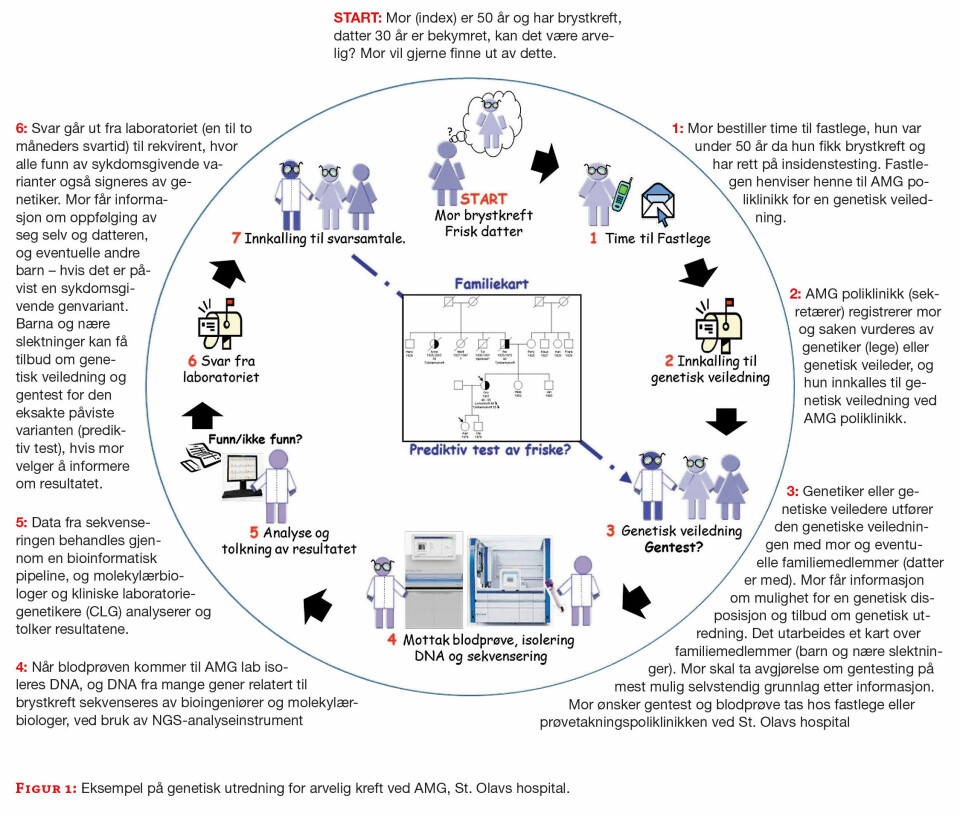
I de tilfeller man ikke kjenner den genetiske årsaken til sykdommen fra før, er det viktig for familie og helsetjenesten å finne en forklaring på sykdomsutviklingen – for eksempel ved medfødt utviklingsavvik. Genetiske undersøkelser kan være med å støtte opp under diagnosen ved syndromutredning, fordi symptomene ofte kan være overlappende og de kan variere i omfang og alvorlighetsgrad. Også for syndromer er det viktig med korrekt diagnose, med tanke på riktig behandling og oppfølging. Det kan også være gjentagelsesrisiko, noe som er viktig å være klar over ved eventuelle nye svangerskap.
Det er i dag flere behandlingsmetoder som differensieres ut fra om årsaken til sykdom skyldes spesifikke genetiske varianter eller ikke. Persontilpasset medisin er noe helsetjenesten vil satse på fremover, og her er både medfødte og ervervede genetiske faktorer av betydning.
For alle som skal rekvirere medisinsk genetiske analyser i Norge, er det tilgjengelig en nettbasert norsk portal (5). Por-talen inneholder oversikt over alle tilgjengelige genetiske analyser innen medisinsk genetikk i Norge. Man kan søke på indikasjon, underindikasjon, gen og analysenavn, og få informasjon om hvem som setter opp de aktuelle analysene. Portalen har tilgjengelig rekvisisjonsskjema fra alle de medisinsk genetiske avdelingene i landet.
Utvikling av sekvenseringsmetoder – fra Sanger til NGS
Sekvenseringsteknologien hadde sin begynnelse på 1970-tallet med introduksjonen av Sanger-sekvensering, som fortsatt er i bruk. Det første humane genomet ble sekvensert med Sanger-sekvensering. Det tok 13 år og ble publisert i 2001 (6). Dette førte til en utvikling av Sanger-sekvensering med kapillærelektroforese, og etter hvert sekvensatorer med flere parallelle kapillærer. I år 2000 ble den første NGS-teknologien lansert (7), men den ble benyttet kun til forskning det første tiåret. De siste ti til femten årene har det vært en rivende utvikling av NGS, og den har fått sin anvendelse i dia-gnostikken. Nå er det mulig å undersøke større deler av genomet til lavere kostnad enn tidligere.
NGS – sekvensering av korte fragmenter (short-read)
Short-read-sekvensering baserer seg på avlesning av korte sekvenser (vanligvis 150 basepar) og er per i dag den ledende NGS-teknologien på markedet, fullstendig dominert av det amerikanske bioteknologifirmaet Illumina. Det er denne teknologien som benyttes i rutinediagnostikken, og i laboratoriet kan man velge å gjøre enten målrettet sekvensering (kun utvalgte områder av genomet sekvenseres), eksomsekvensering (de protein-kodende delene av genomet sekvense-res) eller helgenomsekvensering (hele genomet sekvenseres, inklusiv ikke-proteinkodende deler og mitokondriegeno-met) (8) (Figur 2).
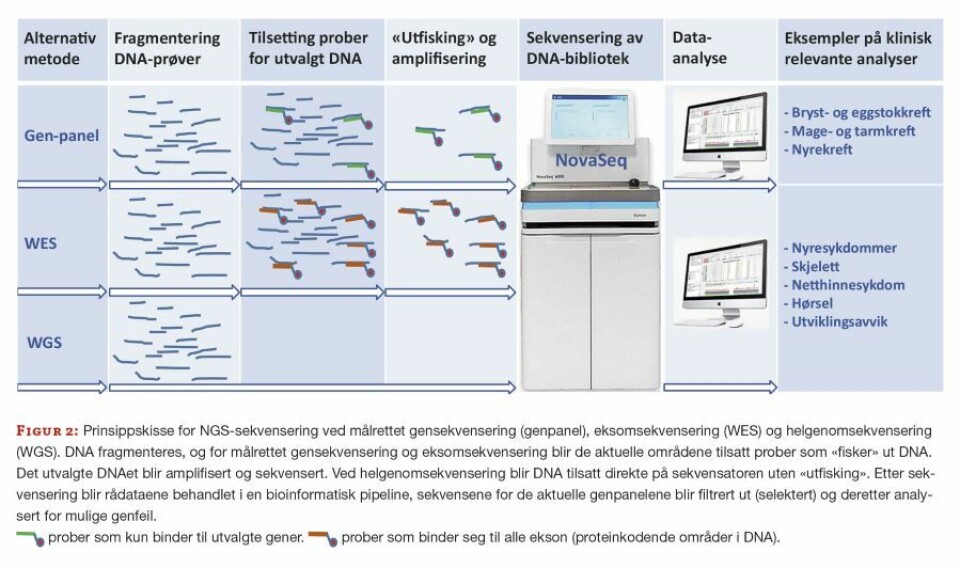
Ved målrettet sekvensering og eksomsekvensering må de delene av genomet som ønskes sekvensert «fiskes ut» på forhånd. En teknisk fordel med helgenomsekvensering er at det krever mindre arbeid å klargjøre prøvene for sekvense-ring (såkalt biblioteksprep, figur 2), siden det ikke skal gjøres en «utfisking» av gener på forhånd, som ved eksom-sekvensering. Ved helgenomsekvensering vil antall ganger hver nukleotide (DNA-byggestein) blir sekvensert være mer likt fordelt gjennom genomet (mer jevn dekning, eller «coverage»). Dette gjør helgenomsekvensering bedre egnet til å analysere større endringer som delesjoner og duplikasjoner av ett eller flere gener, hvilket er en klinisk fordel, fordi mange nyoppståtte utviklingsavvik-syndromer er forårsaket av slike kopitallsvarianter (CNV). Ved å ta i bruk helgenomsekvensering vil man bedre kunne detektere både større og mindre DNA-endringer i samme analyse, og i motsetning til eksomsekvensering vil man også kunne analysere ikke-kodende regioner, som intron og regulatoriske regioner, samt gener i mitokondrielt DNA. Totalt sett vil innføring av helgenomsekvensering kunne føre til at flere får en forklaring på sin genetiske sykdom (9). En av ulempene ved helgenomsekvensering er at det blir mange flere varianter å tolke med hensyn til om de er sykdomsgivende eller ikke. Mange varianter vil ligge i ikke-kodende regioner, og de har vi mindre erfaring med og færre verktøy (software-programmer) tilgjengelige til å hjelpe oss i tolkningen. De vil derfor ofte ende som en variant av usikker klinisk betydning (VUS). Dette problemet vil bli mindre etter hvert som vi får erfaring med tolkning av ikke-kodende varianter, og tolkningsprogrammene vil bli bedre etter hvert som det benyttes kunstig intelli-gens (KI) for å predikere effekt av variantene. Et eksempel på KI som allerede anvendes til tolkning av varianter er prediksjon av proteinstruktur ved hjelp av AlphaFold(10).
Sekvensering av lange fragmenter (long-read)
Long-read-sekvensering er en teknologi som har vakt stor interesse de siste par årene. Metoden representerer tredjege-nereasjonssekvensering. Per i dag domineres markedet av to bioteknologifirmaer som begge tilbyr sekvensatorer og reagenser for long-read-sekvensering, men med forskjellig metodikk: Oxford Nanopore og PacBio. Med long-read-teknologi kan man lese av lengre sekvenser, typisk rundt 10 000 basepar i snitt (11). Dette gjør at man kan lese av og analysere regioner som er krevende med den gjeldende NGS-teknologien, og man kan blant annet avdekke strukturelle varianter, varierende antall triplett-repetisjoner (for eksempel assosiert med Huntingtons sykdom) og om en variant befinner seg på den maternelle eller den paternelle genstrengen. Denne teknologien har derfor et stort potensial for klinisk bruk. Long-read-sekvensering brukes ennå ikke i rutinediagnostikken, men mange laboratorier har tatt tek-nologien i bruk i forskningsprosjekter (12-14). Også ved AMG har vi pågående forskningsprosjekter der vi benytter long-read-teknologi.
Bioinformatisk kompetanse
Moderne sekvenseringsmetoder genererer store datamengder. Dette krever en infrastruktur som kan håndtere analyser av stadig større datasett og som har høy lagringskapasitet. Den stadige økningen i NGS-analyser krever også mer bioinformatisk kompetanse i avdelingene. I tillegg til bioinformatikere, som etablerer og vedlikeholder dataflyt av store datamengder, må også laboratoriepersonell (som bioingeniører) ha kunnskap om den bioinformatiske behandlingen av sekvenseringsdata som foregår før resultatet av gentesten analyseres (for eksempel kvalitetskontroll og diverse filtreringstrinn).
Sekvensering ved AMG
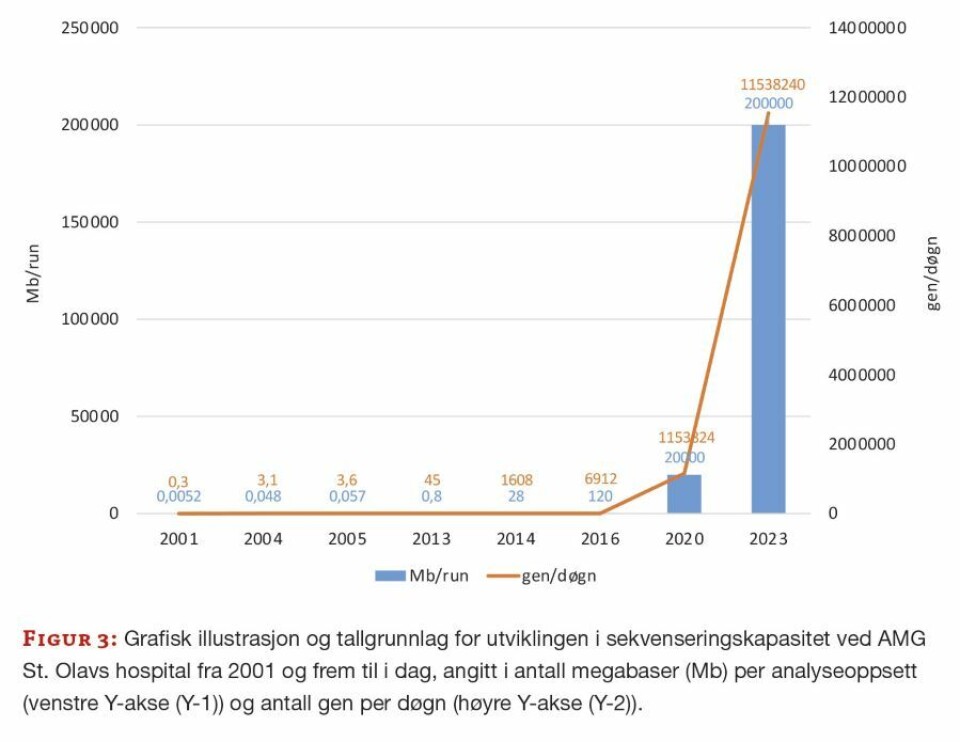
Ved AMG på St Olavs hospital startet vi med vår første sekvensator i 2001, en énkapillærs maskin. Det var da mulig å analysere ett genfragment fra en pasient av gangen, og det tok flere døgn å analysere ett gen. For pasienter som ble utredet for eksempel for arvelig tarmkreft, lå svartiden de første årene på opptil ett år. Etter hvert fikk vi flerkapillær-sekvensatorer; 16 kapillærer i 2005 og 48 kapillærer i 2014. Vår første NGS-maskin fikk vi i 2012, en Roche GS Junior (pyrosekvensering) med kapasitet til å analysere fire gener fra 48 pasienter. Analysen tok totalt en liten uke, og svartiden ble redusert til noen få måneder. Denne teknologien ble snart utdatert, og vi kjøpte vår første Illumina-maskin i 2016, en MiSeq. Siden utviklingen i feltet skjer så raskt, og det blir dyrt å stadig bytte ut sekvensatorene, har vi siden 2020 samarbeidet med kjernefasiliteten for sekvensering ved universitetet (NTNU). Fra 2020 fikk vi tilgang til Illumina NextSeq, og fra 2023 NovaSeq. Vi startet med eksomsekvensering i 2021, og er nå i ferd med å innføre helgenomsekvensering. Vi har som mål å erstatte majoriteten av eksomsekvenseringen med helgenomsekvensering i diagnostikken i løpet av 2024. Utviklingen ved vår avdeling med fokus på kapasitet er illustrert i Figur 3.
Anvendelse av NGS i diagnostikken
Ved AMG benytter vi målrettet sekvensering ved kreftutredning og eksomsekvensering ved syndromutredning. Det betyr at rent laboratorieteknisk behandles alle pasientprøver for utredning av arvelig kreft likt, og vi sekvenserer et utvalg på cirka 80 gener assosiert med arvelig kreft. Når sekvenseringsdataene skal analyseres, analyserer vi derimot kun de genene som har relevans for den aktuelle kliniske problemstillingen. Disse filtreres ut bioinformatisk. Hvis indikasjonen er bryst- og/eller eggstokkreft, analyserer vi for eksempel et panel som består av 21 gener. I noen familier er det mange ulike krefttyper, og dette fører ofte til at vi må re-analysere for å se på et annet panel (for eksempel Mage-tarmkreft på 24 gener). Derfor har vi nylig innført et panel på 50 gener, som dekker de mest vanlige formene for arvelig kreft. Oversikt over alle genpanel kan rekvirentene finne i genetikkportalen (5).
Det samme prinsippet gjelder for syndromutredning: Vi utfører eksomsekvensering for alle pasienter som er henvist for syndromutredning, men analyserer kun de genene som har relevans for den aktuelle kliniske problemstillingen. Her benytter vi genpaneler som er satt sammen av Genomics England PanelApp (15) som utgangspunkt for hvilke gener vi har med i våre genpaneler. Eksempler på paneler som tilbys er Nyresykdommer (259 gener), Netthinnesykdom (201 gener), Hørsel (113 gener), Skjelett (380 gener) og Utviklingsavvik (1973 gener).
Oppsummering
Revolusjonen som har vært innen NGS har medført en dramatisk økning i kapasitet og mye kortere svartid. Dette har kommet pasientene til gode, både i form av kortere ventetid for svar på en genetisk utredning og i form av et bredere analysetilbud.
Da vi begynte med Sanger-sekvensering i 2001, brukte vi en uke på å sekvensere ett gen i en pasientprøve. Nå i 2023 sekvenserer vi på en uke 48 pasientprøver (80 kreftassosierte gener) med NGS til målrettet sekvensering, eller 12 pasi-entprøver (hundre til tusenvis av gener avhengig av fenotypen) med eksomsekvensering. Fra i år er målet å benytte helgenomsekvensering til syndromutredning. Da vil også kopitallsanalyse for alle gener kunne gjøres.
Ny teknologi har skapt behov for en ny type fagfolk og en ny type kompetanse. Tolkning av varianter er blitt et stort fagfelt, og bioinformatikere er blitt en uunnværlig del av staben ved AMG.
























