Bakgrunn
Flere studier antyder at resistensgener stammer fra jordbakterier som produserer antimikrobielle midler eller deler samme miljø som disse produsentene (1, 2). Disse genene har sannsynligvis utviklet seg for å beskytte produsentene mot selvødeleggelse. Det er spekulert i om resistensgener på en eller annen måte har sluppet unna jordbakteriene, og at de etter DNA rekombinasjoner og horisontal overføring er blitt tatt opp av bakterier som har direkte kontakt med mennesker og dyr (2). Bruken av industrielt fremstilte antimikrobielle midler har trolig bidratt sterkt til denne prosessen.
Resistensgener er ofte lokalisert på ekstrakromosomale, selvreplikerende genetiske elementer kalt plasmider eller konjugative elementer kalt transposoner. Resistensplasmider har blitt identifisert i et bredt spekter av bakterier. Multiresistens plasmider er ofte et resultat av rekombinasjoner i plasmidet, integrering av transposoner og/eller integrering av genkassetter i integroner. Alle resistensgenene som er lokalisert på et multiresistens plasmid overføres hvis plasmidet beveger seg over til en annen vert. Dette er mulig dersom plasmidet er et konjugativt plasmid.
Transposoner er overførbare genetiske elementer, også kalt ”hoppegener”, som kan overføres fra et område i genomet til et annet ved en prosess som kalles transposisjon. Genene involvert i transposisjonene kalles transposaser (tnp) og er en del av transposonet (3). tnpA og tnpR er eksempler på gener involvert i transposisjonen.
Integroner er immobile genetiske elementer i seg selv, men kan overføres med plasmider og transposoner. Det mest utbredte integronet er klasse 1 integron som består av to konserverte segmenter; 5’- og 3’ konservert segment, og mellom dem et variabelt område der genkassettene er lokalisert. Det konserverte 5’ segmentet oppstrøms for det variable området, inneholder et integrasegen, intI, som er vist å katalysere setespesifikk rekombinasjon (4). Genkassettene er små mobile strukturer på ca 250-1500 bp, ofte antibiotikaresistensgener, og det er i dag beskrevet mange ulike genkassetter (4, 5).
Mange streptomycinresistensgener er beskrevet fra kliniske bakterieisolater (6, 7). Det integronassosierte aadA genet koder for en 3’(9)-O-adenylyltransferase og finnes blant annet på overførbare elementer som Tn7 og Tn21 (7). Et av de mest utbredte streptomycinresistensgenene blant Gram-negative bakterier, er de koblede resistensgenene strA-strB. Disse genene koder for to enzymer; aminoglykosid 3’’-fosfotransferase og aminoglykosid 6’’-fosfotransferase, som modifiserer streptomycin (6). strA-strB genene medfører streptomycinresistens hos bakterier fra mennesker, dyr, planter og jord (7).
Hos menneske- og dyreassosierte bakterier (E. coli, Salmonella, Pasteurella, Bordetella) finner man ofte strA-strB genene på små ikke-konjugative ”broad-host-range” plasmider som RSF1010 der de ofte er koblet med sulfonamidresistensgenet sul2. I jord- og plantebakterier, for eksempel Pseudomonas sp., Xanthomonas sp., Erwinia amylovora med flere (7), forekommer strA-strB genene som regel som en del av transposon Tn5393, som er lokalisert på store konjugative plasmider eller på kromosomet (7, 8, 9). strA-strB genene er også funnet i fiskebakterien Aeromonas salmonicida (10).
Det er tre kjente sulfonamid resistensgener som koder for antibiotikaresistent dihydropteroat syntase; sul1, sul2 og sul3 (11, 12, 13, 14). sul1 er som oftest lokalisert på 3’ konservert segment hos klasse 1 integroner (11, 14), mens sul2 vanligvis er lokalisert på små plasmider fra IncQ inkompatibilitetsgruppen som for eksempel RSF1010 (11). Sulfonamidresistens i Gram-negative bakterier er i hovedsak sett i forbindelse med plasmider, men kromosomale mutasjoner er også kjent (11).
Det er kjent minst 20 trimetoprimresistensgener (dfr), hvorav de fleste er genkassetter i integroner (15). Det mest utbredte DHFR genet blant Gram-negative bakterier er dfrA1, som ser ut til å spres blant annet med transposon Tn7 (14). Hos trimetoprimresistente kliniske bakterieisolater er Tn7 som regel lokalisert på kromosomet.
Det er kjent 38 forskjellige tetrasyklin, tet gener, og oxytetrasyklin, otr gener (16). Tetrasyklindeterminantene Tet A og Tet B er efflux proteiner og vanligvis assosiert med store, konjugative plasmider (17). Av de 23 efflux proteinene er 21 funnet kun hos Gram-negative bakterier (18). tet efflux gener hos Gram-negative bakterier finnes vanligvis på transposoner (17).
Hensikten med prosjektet var å undersøke om antibiotikaresistente E. coli kunne forekomme i avføringen fra ville dyr, samt å karakterisere eventuelle resistensgener for å kunne vurdere graden av slektskap med tilsvarende gener funnet i patogene bakterier hos mennesker, dyr, planter og i jord.
Materialer og metoder
Bakteriestammer
Totalt ble 139 E. coli isolater fra tarmfloraen hos ville dyr undersøkt i denne studien. Av disse inngikk 137 E. coli isolater i Helseovervåkningsprogrammet for hjortevilt (HOP) i årene 2001-2003 (19). Resultatene fra HOP-studien, deriblant resistensresultatene, er publisert i Acta veterinaria Scandinavica 2005 av Lillehaug et al. (19). I tillegg ble det inkludert to streptomycinresistente stammer (fra bjørn og elg) isolert i forbindelse med andre studier (20). Resistensbestemmelse av E. coli isolatene ble utført med mikrobuljongdilusjonsmetoden VetMICTM (Statens Veterinärmedicinska Anstalt (SVA), Uppsala, Sverige) etter leverandørens anbefalinger. Den antimikrobielle aktiviteten ble målt ved å bestemme ”minimum inhibitory concentration” (MIC); den laveste konsentrasjonen av et antimikrobielt middel som skal til for å hemme veksten av en mikroorganisme. Videre ble konjugeringsforsøk utført for å undersøke resistensgenenes mobilitet.
DNA-teknikker
Total DNA ble isolert ved hjelp av en kokemetode eller ved bruk av Easy-DNATM Kit Version E (Invitrogen™, Carlsbad, California, USA). Det ble benyttet fire metoder for å isolere plasmider. En småskalametode beskrevet av Birnboim og Doly (21), en storskalametode beskrevet av Kado og Liu (22), samt to kommersielt tilgjengelige metoder fra QIAGEN® (Venlo, Nederland); QIAprep® spin miniprep kit og QIAGEN® Plasmid Purification Using Maxi Kits.
Mange resistensgener hos E. coli og andre Gram-negative bakterier er karakterisert og beskrevet og det ble benyttet primere fra kjente sekvenser for å kartlegge relevante resistensgener. PCR ble utført for å detektere resistensgenene sul1, sul2, strA-strB, aadA1, tranposonene Tn1721 og Tn5393, integron klasse 1 og tetrasyklinresistensgenene tetA og tetB.
Hybridiseringanalyser ble benyttet for å påvise tilstedeværelsen av et gen og for å studere hvordan genene var organisert i genomet. Det ble benyttet prober merket in vitro med [a-32P]dCTP (Montebello Diagnostics, Oslo, Norge/Hartmann analytic, Braunschweig, Tyskland).
Sekvenseringsanalyser ble utført for å kartlegge slektskap mellom resistensgener hos E. coli fra ville dyr med tilsvarende genetiske strukturer hos isolater fra patogene bakterier fra mennesker, dyr og planter.
Resultater og diskusjon
Antibiotikaresistens hos ville dyr
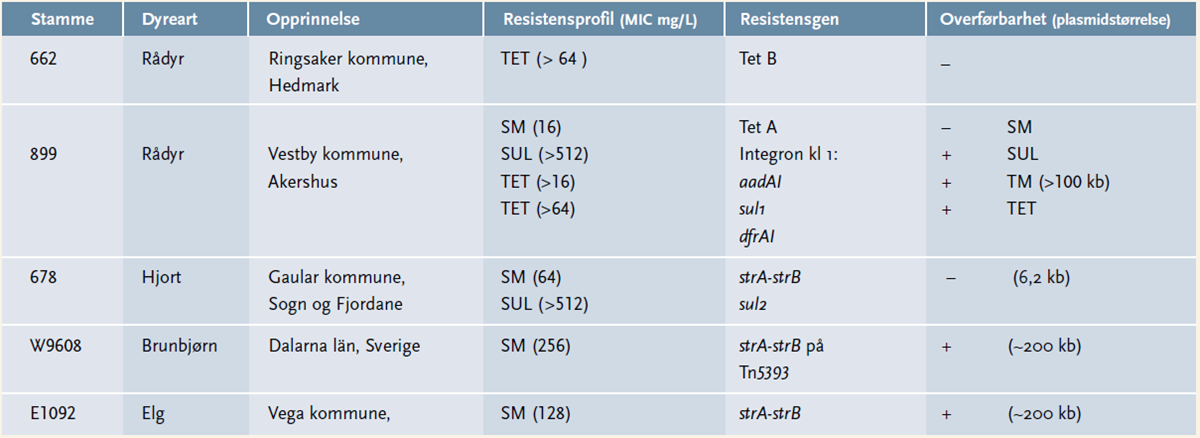
Vi fant i vår studie ut at kun tre av de 137 E.coli isolatene (2,2 %) fra HOP-studien som vi undersøkte, var resistente. Stamme 899 (rådyr) var resistent overfor fire antimikrobielle midler, stamme 678 (hjort) overfor to og stamme 662 (rådyr) overfor ett antimikrobielt middel. W9608 (bjørn) og E1092 (elg), fra andre studier, viste seg å være resistente overfor kun streptomycin. Se tabell 1 for oversikt over resultatene.
Andelen av resistente isolater i denne studien var vesentlig lavere enn for E. coli isolert fra avføringsprøver hos storfe og svin i Norge. Slike bakterier er blitt testet via overvåkningsprogrammet for antibiotikaresistens i Norge NORM/NORM-VET (www.vetinst.no).
Ved seksjon for Vilthelse ved Veterinærinstituttet i Oslo ble det i regi av HOP også samlet inn avføringsprøver fra villrein i jaktsesongen 2003 (19). Det ble isolert 42 E. coli isolater fra 50 avføringsprøver fra forskjellige dyr. Ved sammenlikning med isolatene fra elg, hjort og rådyr, var andelen av resistente isolater fra reinsdyr høyt, hele 24 % (n = 10), hvorav tre var multiresistente. Streptomycinresistens var mest utbredt blant reinsdyrisolatene og det ble videre spekulert i om disse dyrene eksponeres for antimikrobielle midler eller liknende substanser i forbindelse med næringsinntaket (19).
I en fransk undersøkelse ble 341 E. coli isolater fra ulike dyrepopulasjoner undersøkt (23). Disse dyrepopulasjonene hadde ulik grad av kontakt med human sivilisasjon. Det var 18 isolater fra ville dyr fra Antarktis eller Gabon som aldri hadde hatt kontakt med mennesker, 71 fra et område med lav befolkningstetthet (fjellområder i Pyreneene, Frankrike), 61 var ville dyr fra områder med høyere befolkningstetthet (Fontainebleau-skogen i nærheten av Paris, Frankrike), mens 128 var gårdsdyr og 42 var kjæledyr fra Pyreneene i Frankrike (23). Undersøkelsen viste at isolatene fra dyr som levde lengst vekk fra human sivilisasjon var helt fri for antibiotikaresistens. I området med lav befolkningstetthet var 17 % av isolatene resistente mot minst et antimikrobielt middel, og fra områder med høyere befolkningstetthet var 49 % av isolatene resistente. Skurnik et al. (23) fant en kobling mellom tilstedeværelsen av integroner og eksponering overfor human sivilisasjon. Klasse 1 integroner ble kun funnet i E. coli isolater fra dyr som var i nær kontakt med mennesker; gårdsdyr (7 %) og kjæledyr (16 %). Alle integronene fra gårdsdyr var svært like og bar samme genkassett; aadA1, alene eller sammen med dfrA1 eller sat1. Integronene fra kjæledyr viste samme fordeling av genkassetter som ble funnet i normalfloraen hos mennesker i en annen studie av Skurnik et al. (24). I den studien ble det vist at integroner kan persistere i E. coli i den humane normalfloraen uten antimikrobielt seleksjonspress. Skurnik et al. (23) foreslo at integronene var til stede på grunn av en ervervet prosess til miljøet rundt, der et antimikrobielt seleksjonspress har overgått en viss terskel. Disse beskrevne funnene viser at tarmbakterier fra populasjoner av ville dyr som lever nær mennesker bærer en høyere frekvens av antibiotikaresistensgener enn populasjoner som har liten eller ingen kontakt med det menneskelige miljøet.
I vår studie ble det funnet antibiotikaresistente E. coli-isolater fra ville dyr med ulik grad av kontakt med human sivilisasjon i fem isolater. Vi gikk videre med tre av disse isolatene. De blir nå nærmere beskrevet.
Antibiotikaresistent E. coli fra hjort, Gaular kommune, Sogn og Fjordane (stamme 678)
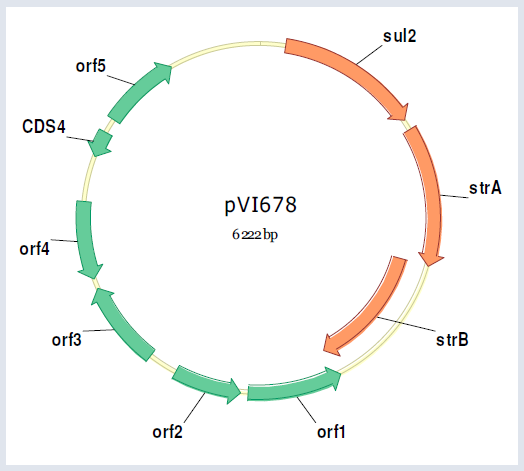
Stamme 678 var resistent mot streptomycin og sulfonamider, og inneholdt resistensgenene sul2 (MIC 128 mg/L) og strA-strB (MIC > 64 mg/L). Resistensgenene var lokalisert ved siden av hverandre på et lite, ikke-overførbart plasmid, se figur 1. Sekvensen er tilgjengelig i GenBank EF090911.
Plasmidet i stamme 678 var på 6222 bp og nukleotidsekvensen var 99,9 % identisk med plasmid p9123 beskrevet og karakterisert av Enne et al. (25). Plasmidet p9123 ble funnet i et klinisk E. coli isolat fra London. Det var tre mutasjoner i posisjon 674 (leseramme sul2), 2930 og 3151 (begge i leserammen til orf1) sammenliknet med p9123. Kun den siste mutasjonen førte til endring av aminosyre fra asparaginsyre til glysin.
p9123 er et ”fitness-plasmid” som forkorter generasjonstiden til den bakterien det er en del av (25). Både dette og liknende plasmider er meget utbredt blant enteriske bakterier, sannsynligvis på grunn av denne egenskapen. Funn av det samme plasmidet i tarmfloraen hos ville dyr viser at resistensplasmidet opprettholdes uten seleksjonspress fra antibiotikabruk.
Antibiotikaresistent E. coli fra brunbjørn, Dalarne län, Sverige (stamme W9608)
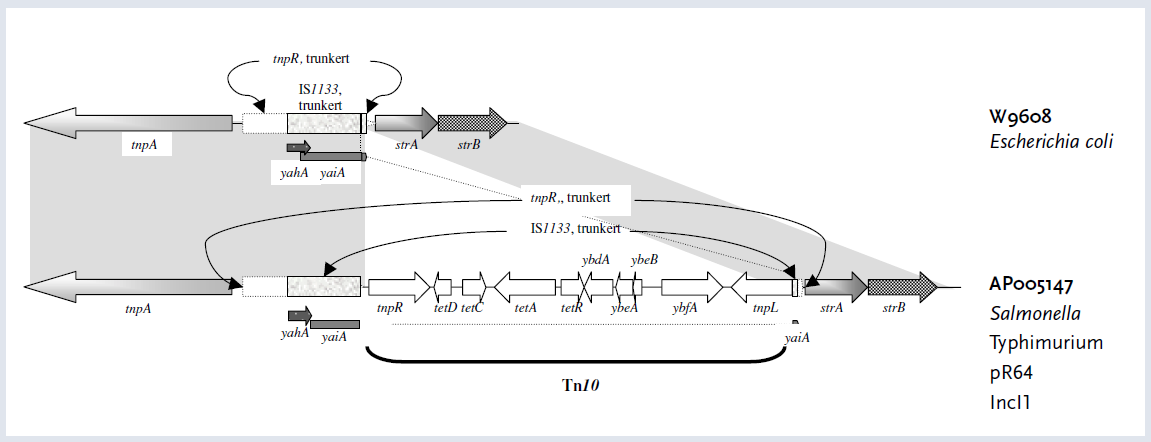
Stamme W9608 inneholdt streptomycinresistensgenene strA-strB lokalisert på transposonet Tn5393. Tn5393 var lokalisert på et stort (ca 200 kb) overførbart plasmid, totalt 9712 bp av plasmidet ble sekvensert. Sekvensen er tilgjengelig i GenBank EF108308. Tn5393 i stamme W9608 inneholdt et trunkert tnpR gen, et trunkert IS1133 element (IS – insertion sequence) og rester av ”inverted repeat” sekvenser fra transposonet Tn10. BLASTN søk viste at dette området var mest beslektet med et område på et Salmonella Typhimurium plasmid (AP005147). Dette plasmidet har et transposon Tn10 innsatt i IS1133 elementet på et Tn5393 transposon, se figur 2. Denne stammen inneholder plasmidet pR64, som er et stort plasmid (120 kb) som tilhører IncI1 inkompatibilitetsgruppen.
Dette er første gang et Tn5393 er funnet hos E. coli fra pattedyr. Tn5393 er et 6,7 kb langt transposon som er i stand til å erverve IS elementer for å øke ekspresjonen av streptomycinresistens i ulike slekter av bakterier (26).
Området nedstrøms og oppstrøms for IS1133 hos S. Typhimurium (AP005147) og W9608 (E. coli) ble sammenliknet og viste tilnærmet 100 % homologi for disse stammene. Dette kan indikere at IS1133 elementet har blitt satt inn i tnpR og at transposonet Tn10, et klasse I transposon, har vært tilstede i W9608 for så å bli deletert vekk fra plasmidet og etterlate seg flankerende områder som fortsatt finnes i stammen.
Det finnes ikke bevis på nylig overføring av strA-strB genene mellom menneske-/dyreassosierte og planteassosierte bakteriegrupper, men det finnes sterke indikasjoner for at strA-strB genene som finnes på små ikke-konjugative sulfonamid- og streptomycinresistente plasmider opprinnelig var linket til Tn5393 sekvensen (7). Dette transposonet kan ha utviklet seg i en jordbakterie som et resultat av naturlig streptomycinseleksjon fra streptomycinproduserende bakterier (7). Oppdagelsen av Tn5393 sekvenser i flere bakterier fra ulike geografiske områder indikerer at dette transposonet er del av en ”pool” av gener som er tilgjengelig for et bredt spekter av organismer (26). Streptomycin brukes lite til behandling av infeksjoner i humanmedisinen i dag, slik at persistensen av strA-strB i kliniske bakteriepopulasjoner tyder på at andre faktorer enn direkte antimikrobiell seleksjon kan være involvert i vedlikeholdet av disse genene (26).
Antibiotikaresistent E. coli fra rådyr, Vestby kommune, Akershus (stamme 899)
Stamme 899 inneholdt et integron klasse 1 med genkassettene sul1, dfrA1 og aadA1 som medførte resistens mot sulfonamider, trimetoprim og streptomycin. aadA1 ble uttrykt lavgradig på grunn av svak promoter. I tillegg inneholdt stammen tetrasyklinresistensgenet tetA lokalisert på et trunkert transposon Tn1721. Disse områdene var lokalisert på et stort (ca 200 kb) overførbart plasmid.
Det var ingen nukleotidsekvenser ved BLASTN søk som hadde nøyaktig samme organisering av klasse 1 integronet og flankerende DNA, men det var meget stor grad av homologi mellom området oppstrøms for tetA(A) og flere publiserte sekvenser. Området nedstrøms for tetR(A) ble sekvensert og et oriV, som er et incP vegetative ”origin of replication”, ble påvist 353 bp nedstrøms for tetR(A). Sekvensen til området oppstrøms for tetA(A) var blant annet 100 % homologt med et område på IncP-1a plasmid; pTB11 (AJ744860) og plasmid pRSB101 (AJ698352), begge isolert fra renseanlegg for avløpsvann, og plasmid pKBB958 (AM183165) isolert fra Bordetella bronchiseptica fra svin. Tennstedt et al. (27) viste at bakterier med IncP-1 plasmider frigjøres i miljøet med avløpsvannet fra slike renseanlegg. Tetrasyklinresistensområdet på plasmidet pTB11 er nesten identisk med en indre del av Tn1721 og det er sannsynlig at dette DNA-området representerer den sentrale kjernen av Tn1721 (27).
Avslutning
De tre E. coli isolatene som ble valgt ut for videre genetiske studier viste seg å ha forskjellig organisering av resistensgenene. Stamme 678 hadde strA-strB og sul2 genene lokalisert ved siden av hverandre på et lite ikke-overførbart plasmid. Stamme W9608 inneholdt et stort overførbart plasmid der strA-strB genene var lokalisert på transposon Tn5393 som inneholdt IS elementet IS1133. Stamme 899 inneholdt et klasse 1 integron med genkassettene dfrA1, aadA1 og sul1. I tillegg inneholdt stammen tetrasyklinresistensdeterminanten Tet A som var del av et trunkert transposon Tn1721. Disse resistensgenene var lokalisert på et stort overførbart plasmid.
Skurnik et al. (23) fant at klasse 1 integroner kun ble detektert i E. coli isolater isolert fra avføringen hos dyr som hadde vært i nær kontakt med mennesker eller menneskelig aktivitet. E. coli isolatet fra stamme 899, med klasse 1 integron, ble isolert fra et rådyr i Vestby kommune i Akershus fylke. Dette området ligger cirka fire mil utenfor Oslo og er relativt tett befolket. Det er imidlertid også vist at klasse 1 integroner kan forekomme i miljøer langt fra mennesker hvor antimikrobielle midler aldri brukes (23, 28). Det er blant annet vist at et E. coli isolat fra avføringen fra et reinsdyr som levde i et fjellområde i midt-Norge (Forelhogna, 900-1200 moh), inneholdt et klasse 1 integron med genkassetten aadA1 og et intron klasse II (28). Dette kan indikere at klasse 1 integroner er mer vanlig i naturen enn man først trodde, og at de trolig har eksistert siden før antimikrobielle midler ble framstilt industrielt og tatt i bruk for å behandle infeksjonssykdommer (28).
I de to andre stammene (678 og W9608) ble E. coli isolert fra avføringen hos dyr som har oppholdt seg i lite befolkete områder, blant annet Gaular kommune i Sogn og Fjordane (678) og Dalarna län i Sverige (W9608). I disse to stammene ble strA-strB genene funnet og det kan indikere at resistensgenene kan ha blitt overført fra streptomycinproduserende bakterier i jorda.
Likhetstrekk mellom resistensgener og organiseringen av dem er funnet mellom enterobakterier og plante- og jordbakterier. IncP-1a plasmider bidrar til ”poolen” av horisontalt overførbare genetiske elementer som er assosiert med kloakkbakterier. Flere hot-spots for horisontal genoverføring av genetisk materiale er oppdaget i miljøet. Blant disse er renseanlegg for avløpsvann spesielt viktige siden de mottar spillvann/kloakk som inneholder vesentlige mengder antimikrobielle midler og resistente bakterier fra sykehus, privat hushold, industri og landbruk (27, 29).
Denne studien har vist at viltlevende dyr, som ikke har vært behandlet med antimikrobielle midler, også kan ha antibiotikaresistente bakterier i sin mikrobielle tarmflora. Resistensgener funnet hos disse bakteriene tilsvarer resistensgener som er vanlig forekommende hos human- og dyrepatogene bakterier. Organiseringen av resistensgenene hos bakterier fra ville dyr er noe annerledes enn hos human- og dyrepatogene bakterier. Det er grunn til å anta at antibiotikaresistens er en ”naturlig egenskap” hos enkelte sjeldne E. coli stammer – og at det ikke utelukkende er en konsekvens av menneskelig bruk av antimikrobielle midler.